与专家对话:设计用于高通量 CRISPR 筛选的 sgRNA 文库
以下是 Twist 为《基因工程新闻》编写的电子书的摘录:《CRISPR 筛选的变化格局:最新 CRISPR 筛选方法和技术指南》。您可以在此处下载完整的电子书。
即使对于经验丰富的研究人员来说,设计 sgRNA 文库也可能是一项艰巨的任务。数千个文库中的每个指南都需要仔细考虑,以确保其靶基因的保真度。而且,准确性并不总是等同于有效性——大约 20% 的 CRISPR 诱变会导致框内突变,其中许多突变不太可能影响蛋白质功能1.自 CRISPR 首次应用于哺乳动物细胞的基因编辑以来的十年中,设计 sgRNA 文库的指南不断发展,以应对新的见解和新技术。随着这种演变,对定制 CRISPR sgRNA 文库的需求不断增长,这些文库经过定制,旨在最大限度地提高效率,同时充分利用 CRISPR 筛选的发现能力。
在这里,我们与 Twist 的两位 CRISPR 专家坐下来讨论 sgRNA 文库设计的基础知识,以及他们对考虑定制方法的人有什么建议。Julian Jude 博士是著名的 CRISPR 专家,也是用于对 sgRNA 设计进行评分的维也纳生物活性 CRISPR (VBC) 算法的合著者1.十年来,Julian 一直在帮助研究人员改进 CRISPR 筛选。与 Julian 一起的是 Twist Bioscience 的首席技术官 Siyuan Chen 博士。
研究人员在设计 sgRNA 文库时应该从哪里开始?
思源:为您的实验设计最佳的 sgRNA 文库在一定程度上取决于您的筛选目标。用于 CRISPRi 筛选的完美 sgRNA 与用于经典基因敲除筛选的完美 sgRNA 不同,应插入与您的递送方法相匹配的骨架中。因此,重要的是要从一个建立在通用规则之上的立场开始,但最终会考虑您的特定需求。
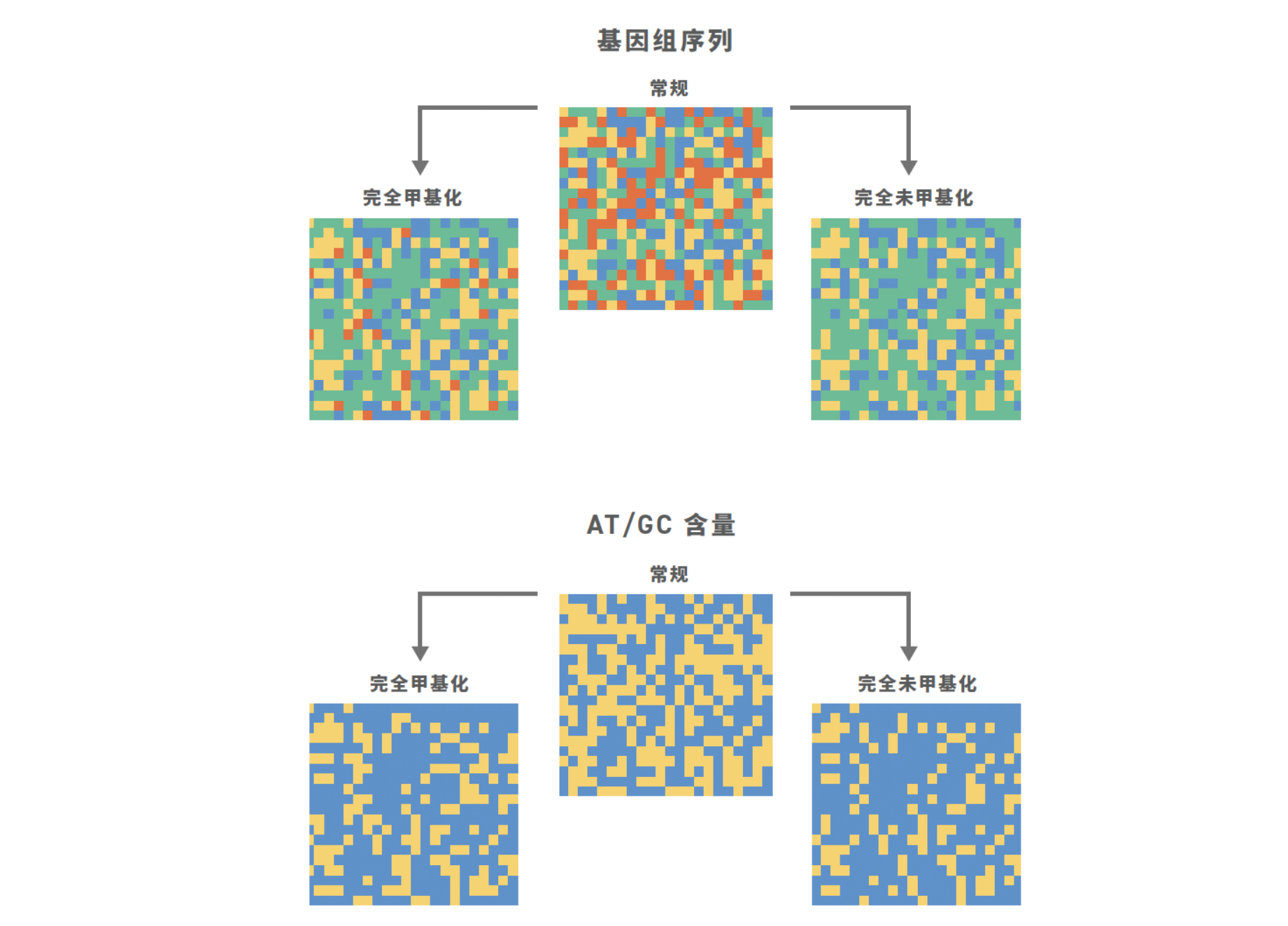
朱利安:正如你所说,一个好的 sgRNA 文库是经过精心设计的,以满足特定需求。但是有一些普遍性是应该考虑的。有许多优秀的资源可以帮助感兴趣的研究人员了解 sgRNA 设计的基本规则。简而言之,sgRNA 应与非靶标 DNA 序列具有最小的互补性,GC 含量在 40% 到 80% 之间,并且 sgRNA 的间隔区 RNA 部分的长度应延长约 17 至 24 个核苷酸(确切长度取决于您选择使用的相关 Cas 蛋白)。
确定要使用的合适 CRISPR 相关 (Cas) 蛋白是一个关键步骤。在这个阶段,已经为基因组筛选开发了许多不同类型的核酸酶。要选择适合您的那一种,需要分析靶基因附近的可用 PAM 序列,并考虑您需要 Cas 蛋白进行哪种类型的扰动。如果您需要这方面的指导,我建议您查看 John Doench 2019 年对 CRISPR 技术的评论2,其中包括各种 Cas 蛋白、其功能及其相关 PAM 序列的良好概述。
可用于 sgRNA 设计的最新和最好的工具是什么?
朱利安:如果您的目标是进行基因敲除筛选,则有一些方法可以优化 sgRNA 以增加实现靶向敲除的可能性,或者至少诱导功能丧失的框内突变。
我最近是奥地利维也纳生物中心研究团队的一员,他们开发了维也纳生物活性 CRISPR 评分 (VBC),这是一种高性能 sgRNA 预测工具,可帮助研究人员选择在哺乳动物细胞中可靠地产生功能丧失等位基因的 sgRNA1.在开发该工具时,我们评估了最有可能成功导致功能丧失突变的 sgRNA 的质量。我们的分析显示,旨在靶向高度保守氨基酸片段的 sgRNA 更可能导致蛋白质功能丧失,在 7 个氨基酸(21 个碱基对)的片段中观察到最佳结果。同样,靶向与蛋白质核心中疏水结构域相对应的 DNA 序列的 sgRNA 更有可能表现良好。
方框 1:CRISPR sgRNA 设计的一般注意事项
- GC 含量在 40% 到 80% 之间,以确保 sgRNA 与目标 DNA 之间的强结合。
- 确保在 sgRNA 设计中限制发夹和聚合酶终止序列等二级结构。这些结构会影响克隆效率和指南的转录。
- 删除 polyA 网站,如果您使用病毒式传播,这些网站可能会破坏包装。
- 最大限度地减少错配,尤其是在 PAM 上游的前 10 个碱基内。在间隔序列的后半部分可以容忍错配,但靠近 PAM 位点的错配可能会破坏结合和随后的编辑。
- 将 sgRNA 中的间隔序列长度与所使用的 Cas 蛋白相匹配。这两者之间的不一致会严重降低编辑效率。
- 研究并选择最适合您需求的 Cas 蛋白。Cas 蛋白在识别的 PAM 序列、执行的切割类型以及影响编辑性能的多个其他因素方面有所不同。
- 确保 sgRNA 启动子不会与其他启动子重叠冲突,例如经常用于转录选择抗性基因的 EF-1a 启动子。与该启动子重叠导致 sgRNA 转录效率降低。
此框的见解来自最近的几项研究1-7.这些见解来自专注于哺乳动物细胞的研究,在其他系统中可能并不成立。
我们的工作中强调并在 VBC 评分中捕获的是,当我们不仅考虑 sgRNA 特异性,而且根据基因和蛋白质结构选择指南时,sgRNA 文库更有可能实现敲除。[参见方框 1 和 2 了解本研究得出的 sgRNA 的一般注意事项列表。
思源:对于旨在通过 CRISPRi、CRISPRa、CRISPRon 或 CRISPRoff 调节基因表达的实验,存在一些限制限制可供选择的 sgRNA 数量。例如,研究表明,CRISPRi sgRNA 在靶向转录起始位点下游 75 个碱基对窗口内的序列时最有效。CRISPRoff 的这个窗口被拓宽,使得靶向转录起始位点上游或下游 500 个碱基对内的序列的 sgRNA 在转录抑制方面似乎最有效3.
朱利安:此外,由于单细胞 CRISPR 筛选可以使用敲除或表观遗传扰动方法,因此还必须设计这些 sgRNA 文库以确保每个 sgRNA 都忠实地与其扰动和随后的转录组谱相关联。单细胞 CRISPR 筛选一直具有挑战性,因为功能性 sgRNA 不能被多聚腺苷酸化,但许多 RNA 测序方法依赖于多聚腺苷酸化转录物的捕获。
已经开发了通过将条形码插入质粒选择基因下游 3' 长的末端重复部分来克服这一障碍的方法。虽然这种方法有效,但也有一些局限性,例如在克隆步骤中由于模板切换而导致条形码与 sgRNA 解离(多项研究表明,这可能会影响多达 50% 的质粒)4-6.
因此,考虑 sgRNA 在骨架中的插入位置以及在测序时如何检测它非常重要。CROP-seq 和直接捕获 Peturb-seq 等方法提供了替代方法。[有关最新单细胞 CRISPR 筛选技术的深入分析,请参阅我们电子书的第 7 章]有几种工具可以帮助研究人员设计 sgRNA,包括前面描述的 VBC 评分。这些工具已收集在一个有用的 github 存储库中。
方框 2:特定于上下文的注意事项
基因敲除 sgRNA 设计注意事项
- 靶向导向保守氨基酸序列(理想情况下长度为 7aa)。这些序列很可能是保守的,因为它们对蛋白质功能很重要,因此这些区域的破坏更有可能有效。
- 靶向蛋白质核心中的疏水结构域。目前尚不清楚为什么编辑核心疏水结构域更有可能破坏蛋白质功能,但实验证据表明这是一种有效的靶向方法。
- 靶向较大的外显子以避免选择性剪接,因为尽管编辑成功,但这可以规避基因敲除。
- 不要靶向蛋白质编码序列末端附近的区域,因为尽管有轻微的截断,但一些蛋白质可能仍然表达和功能。
- 避免将位置定位到备用起始站点附近。与替代剪接位点一样,替代起始位点可以补偿标准转录本的丢失,以防止基因敲除。
- 避免使用经常包含多态性的位置。即使您的 sgRNA 被设计为完美匹配您的靶序列,靶序列中的多态性也可能导致意外的错配和编辑效率降低。
- 确保 sgRNA 启动子不会与其他启动子重叠冲突,例如经常用于转录选择抗性基因的 EF-1a 启动子。与该启动子重叠导致 sgRNA 转录效率降低。
CRISPRi/a/开/关 sgRNA 设计注意事项
- 由于您需要靶向转录起始位点/CpG 岛,因此可以使用有限的 sgRNA 选项
- 对于 CRISPRi sgRNA,理想的靶标窗口是 TSS 下游 +25 至 +75 nts,基于显示该窗口内基因表达最佳破坏的平铺阵列。
- 对于 CRISPRa:TSS 上游的 150-75 个核苷酸提供了最佳激活。
- CRISPRoff 和 CRISPRon sgRNA 在靶向以转录起始位点为中心的 1 kb 窗口时最有效。该区域的甲基化可能会破坏关键转录因子的结合和聚合酶的募集。
此框的见解来自最近的几项研究1-7.这些见解来自专注于哺乳动物细胞的研究,在其他系统中可能并不成立。
有很多预制的 CRISPR sgRNA 文库可用。研究人员应该使用这些还是考虑定制设计的库?
思源:对于对设计自己的 sgRNA 文库持谨慎态度或不确定用于 CRISPR 筛选的正确骨架的研究人员有两种选择:使用现成的文库进行一般筛选,或者获得设计自定义 sgRNA 文库的帮助。
对于预算有限并希望使用其他人已验证的库的研究人员来说,前者可能是一个不错的选择。这些库通常已准备就绪,这意味着制作和部署它们所需的等待时间很短。但是,此类库有一些明显的缺点。CRISPR 领域的发展速度如此之快,以至于任何静态 sgRNA 文库在使用时都可能已经过时。预制文库也无法满足研究人员的特定需求 — 文库旨在满足多个潜在的研究目标,并且可能会对特定感兴趣的领域提供过多或过少的覆盖。
定制 sgRNA 文库具有许多优势。定制 sgRNA 文库概念的固有特点是灵活性,这种灵活性对于使用一些较新的 CRISPR 技术的筛选特别有用。
朱利安:一个很好的例子是在 prime-editing 领域。Prime 编辑在大规模筛选中具有精确扰动的巨大潜力,但这样做需要非常长的 sgRNA 序列文库。引物编辑 sgRNA (pegRNA) 必然比 sgRNA 长,因为它包括一个 sgRNA 和一个额外的 30+ 核苷酸,这些核苷酸编码引物结合位点和修复模板8.为了促进这一点,必须使用长寡核苷酸合成精确构建 pegRNA 文库。很少有公司能做到这一点,但 Twist Bioscience 就是其中之一。
定制文库还使您能够使用质粒骨架、tracrRNA、条形码的位置和其他可能有助于您改进筛选的功能,而预制文库通常具有这些固定的功能。
“不要害怕寻求帮助,要有创造力”
我们还看到研究人员将预制库与定制解决方案相结合。大规模筛查会导致成百上千次需要跟进的扰动。最有效的方法之一是利用自定义向导文库进行集中的二次筛选,该文库旨在靶向初级全基因组筛选中鉴定的所有基因。因此,研究人员可以从定制 sgRNA 文库和预制文库中获得大量使用。这取决于您的时间表、资源和需求。但是,无论您选择什么库,您都需要确保它是统一的,并且创建时错误率较低。
研究人员在哪里可以获得均匀、低误差的 CRISPR sgRNA 文库?
思源: Twist 生物科学。Twist 的硅基寡核苷酸合成平台能够快速生成低错误、高度均一的寡核苷酸,然后将其转化为 sgRNA 文库。这些均匀性和低误差的品质对于任何 CRISPR 筛选实验都极具吸引力。
朱利安:我同意,统一是关键。Uniformity 是每个参考线相对于库的其余部分出现的频率的描述。高度均匀的库将平等地表示每个参考线,而低均匀性的元器件库可能包含一些精选参考线的过度表示。筛选时,一致性很重要,因为不均匀的文库会导致代表性不足的指南的感知损失或效果减弱。换句话说,非均匀文库会降低筛选敏感性,并可能导致结果有偏差。
为了补偿不均匀性,筛选需要更多的细胞,以确保可以检测到代表性不足的向导的影响。因此,均一文库能够以更少的细胞成功筛选——这是处理数量可能受限的原代细胞系时的一个重要功能。这就是为什么在设计 sgRNA 文库时,确保选择提供高度均一的寡核苷酸池的供应商非常重要。我们在关于均匀性重要性的白皮书中对此进行了更多介绍。
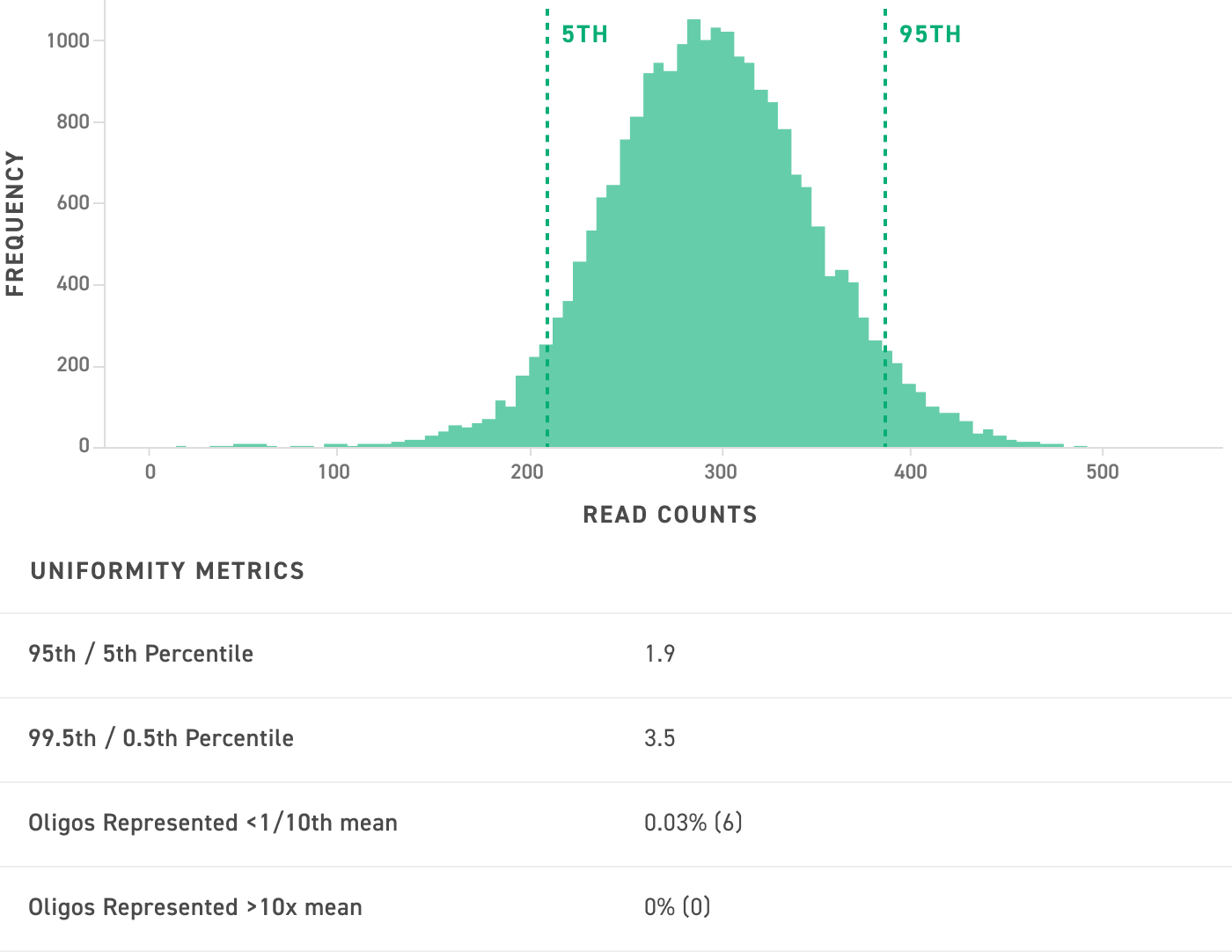
性。相应的表显示了此池的一致性指标。Twist 寡核苷酸合成无偏倚,具有高均一性和完整的寡核苷酸代表性。
思源:错误率同样重要。CRISPR 系统旨在根据 sgRNA 的间隔区部分精确靶向 DNA 序列。sgRNA 的后 10 个核苷酸的错配在一定程度上是可以容忍的;然而,不匹配会增加脱靶效应的可能性。因此,sgRNA 生成过程中的错误会影响 sgRNA 特异性。还值得注意的是,对于使用模板进行修复的应用程序(例如 prime editing),模板编码必须准确无误,这一点至关重要。
借助 Twist Bioscience 的寡核苷酸池,研究人员可以设计定制文库,然后将其克隆到您选择的载体中。如果需要,Twist 的 CRISPR 专家还可以提供有关文库设计、载体和 NGS 策略的一些指导。
sgRNA 设计是否有任何最终考虑因素?
朱利安:我认为指出 sgRNA 设计中一个经常被忽视的方面也很重要:需要保护您的实验免受引物结合位点污染。
NGS 测序 sgRNA 是一种快速简单的方法,用于评估筛选结束时存在哪些 sgRNA。这个过程的关键是具有独特的引物结合位点,使您能够将扩增限制在筛选中的 sgRNA,而不是可能偷偷进入的任何其他 sgRNA 表达载体。在经常使用 CRISPR 的实验室中,引物结合位点污染很容易发生。从质粒中扩增比从基因组 DNA 中扩增要容易得多,因此大多数 NGS 读数都来自污染载体。
一个经常被忽视的问题:引物结合位点污染
发生这种类型的污染的原因有很多。通常,实验室使用位于用于促进 sgRNA 转录的 U6 启动子内的引物结合位点。例如,如果您想在进行筛选之前在细胞系中进行敲除,则很容易重复使用相同的 CRISPR 质粒构建体,包括相同的 U6 启动子,并意外地将其插入引物结合位点。
为了保护您的实验,考虑添加筛选文库独有的引物结合位点非常重要。一种方法是在筛选文库质粒中插入独特的引物结合位点。
最后还有什么鼓励研究人员进入筛选领域的建议吗?
思源:不要害怕寻求帮助并发挥创造力。很多人只想遵循以前做过的事情。但是,有一些创造性的方法来执行筛选,可以帮助改进您的实验。而且,在 Twist Bioscience 等地方,总有专家可以提供帮助。
References
- Michlits, Georg, et al. “Multilayered VBC Score Predicts SgRNAs That Efficiently Generate Loss-of-Function Alleles.” Nature Methods, vol. 17, no. 7, 8 June 2020, pp. 708–716, 10.1038/s41592-020-0850-8.
- Doench, John G. “Am I Ready for CRISPR? A User’s Guide to Genetic Screens.” Nature Reviews Genetics, vol. 19, no. 2, 4 Dec. 2017, pp. 67–80, 10.1038/nrg.2017.97.
- Nuñez, James K., et al. “Genome-Wide Programmable Transcriptional Memory by CRISPR-Based Epigenome Editing.” Cell, vol. 0, no.0, 9 Apr. 2021, www.cell.com/cell/fulltext/S0092-8674(21)00353-6, 10.1016/j.cell.2021.03.025.
- Datlinger, Paul, et al. “Pooled CRISPR Screening with Single-Cell Transcriptome Readout.” Nature Methods, vol. 14, no. 3, 18 Jan. 2017, pp. 297–301, 10.1038/nmeth.4177.
- Hill, Andrew J., et al. "On the Design of CRISPR-Based Single Cell Molecular Screens." 29 Jan. 2018, 10.1101/254334.
- Xie, Shiqi, et al. “Frequent SgRNA-Barcode Recombination in SingleCell Perturbation Assays.” PLOS ONE, vol. 13, no. 6, 6 June 2018, p.e0198635, 10.1371/journal.pone.0198635.
- Mohr, Stephanie E., et al. “CRISPR Guide RNA Design for Research Applications.” The FEBS Journal, vol. 283, no. 17, 22 June 2016, pp. 3232–3238, www.ncbi.nlm.nih.gov/pmc/articles/PMC5014588/, 10.1111/febs.13777.
- Anzalone, Andrew V et al. “Search-and-replace genome editing without double-strand breaks or donor DNA.” Nature vol. 576,7785 (2019): 149-157. doi:10.1038/s41586-019-1711-4
原英文链接
推荐文章
-
Twist推出的多基因片段库(Multiplexed Gene Fragments)首次突破300 bp的传统合成极限,实现500 bp长片段的高通量合成。
-
低起始量样本的靶向甲基化测序通常存在相当大的挑战,因为未甲基化胞嘧啶的转化降低了基因组测序的复杂度。Twist 的甲基化检测系统通过组合探针的能力(可以实现不同级别的优化过滤严谨度)有效地克服了这些障碍,同时还引入了甲基化促进剂,这是一种定制的阻断剂,通过减少脱靶来产生协同作用,以提高系统性能。这两种优化都极大地提高了甲基化检测组合观测到的测序性能,促进了 Twist 甲基化检测系统可支持液体活检等高灵敏度应用的改进。...
-
专家视角|Julian Jude谈功能基因组学的新时代 市场部 Twist Bioscience 拓唯思特 2025年02月18日 17:30 北京 当下正是功能基因组学研究的黄金时代。CRISPR技术工具库的持续扩展,使研究者能以更复杂、更大规模的方式操控基因组;人工智能(AI)助力多模态数据分析,不仅能优化功能筛选设计,还能从中挖掘更深层的生物学意义。如今,功能基因组学研究已迈入规模化与高效化并行的新纪元。这些划时代的进步,离不开合成DNA制备技术的同步革新——这一常被忽视的技术,实为现代分子生物学的基石。 近期,我们与Julian Jude博士 深入探讨了过去十年合成DNA技术的演进,以及它如何推动当今功能基因组学的蓬勃发展。 专家简介 Julian是功能基因组学领域的资深专家,拥有20余篇学术论文及多项专利申请。在攻读博士及博士后期间,他开发了用于靶点识别的新型功能基因组学工具,主导了70余项全基因组shRNA与CRISPR筛选项目,并参与创建了CRISPR单导RNA(sgRNA)设计评分...